Antes del 2010 Synthia no era nada, no existía. Sin embargo, ahora mostraba las características de lo que conocemos como “vivo”. Su existencia no era resultado de la evolución de otro microorganismo, sino del esfuerzo de más de una veintena de investigadores y de millones de dólares invertidos a una serie de proyectos de genética. Mycoplasma laboratorium, como se llamó al organismo, es una cepa de una bacteria sin pared celular creada artificialmente.
Su producción fue toda una odisea que llevó a un equipo formado por algunos de los mejores genetistas del planeta a inventar nuevas técnicas para poder realizar los experimentos. La idea general del proyecto era estudiar cuál era el número de genes mínimo necesario para crear un organismo vivo, es decir, que pudiese alimentarse, reproducirse y relacionarse con el medio. Por ello, partieron de los organismos con el genoma más pequeño que se conocían en la época y estudiaron los genes que no eran esenciales para mantenerlos con vida.
Uno de los organismos, Mycoplasma genitalium parecía el candidato ideal, ya que es capaz de vivir con poco más de 500 genes en su genoma, de 580.070 pares de bases -lo que comúnmente se conocen como “letras” del ADN-.
Como referencia, los humanos tenemos unos 20.000 genes que codifican proteínas (las últimas cifras indican 63.494 en total, contando genes no codificantes) y más de 6 mil millones de pares de bases.
La creación de la vida sintética
M. genitalium debe su nombre a que se trata de una bacteria que fue aislada por primera vez en 1981 a partir de muestras del tracto urogenital. La bacteria es capaz de infectar el tracto urinario y genital, tanto de hombres y mujeres, y se trasmite, generalmente, por contacto sexual. En la actualidad, el aumento en el número de casos es de gran preocupación, ya que provoca dolor, problemas en la micción, sangrado, e incluso puede llevar a provocar partos prematuros. Pero en un espacio confinado como el laboratorio tenían otros problemas con este microorganismo. M. genitalium crece muy lentamente, y el parsimonioso ritmo de esta bacteria chocaba frontalmente con la velocidad a la que se han de realizar los experimentos. Por ello, decidieron sustituirlo por M. Mycoides, con un genoma de casi el doble de tamaño.
Para materializar el genoma completo del organismo comenzaron a ensamblar fragmentos de ADN de entre 5000 y 7000 pares de bases como si se tratase de piezas encajables. Durante el proceso, aprovecharon para introducir ciertas “marcas de agua” que indicaban que el genoma era artificial, así como inscripciones en el código genético. Una vez tenían el genoma completo, trataron de insertarlo en el genoma en otro miembro de la familia Mycoplasma, M. capricolum al que le habían extraído completamente el ADN. Llevó varios intentos, las células se resistían a aceptar este ADN foráneo, pero al final observaron un punto azul en una de las colonias bacterianas que significaba que había comenzado una nueva vida. Es decir, consiguieron que esta célula se transformase en la primera bacteria viva que había sido diseñada en un ordenador.
Algunos genetistas de renombre como George Church, de Harvard, afirmaron que no se podía tildar a este organismo de sintético, ya que se trataba de una reconstrucción de otro microorganismo. Ahora bien, tras esta primera prueba, necesaria para la creación y el perfeccionamiento de la tecnología, en 2016 presentaron una versión con únicamente 473 genes, a la que denominaron JCVI-syn3.0. Este microorganismo tiene un genoma menor que la de cualquier célula que se replique de forma independiente en la naturaleza y sí está reconocido como la primera bacteria completamente sintética.
La familia sintética crece
Aunque JCVI-syn3.0 es la primera bacteria sintética, no es el primer organismo artificial. 13 años antes, en 2003, el Premio Nobel Hamilton O. Smith y el investigador Clyde A. Hutchison sintetizaron el genoma de un virus bacteriófago -que ataca a bacterias- denominado ΦX174. Sin embargo, este organismo tiene únicamente 5386 pares de bases, 100 veces menos que JCI-syn3.0, y solo se puede reproducir infectando bacterias, por lo que el salto cualitativo es notable.
Desde entonces, los avances en las herramientas de edición genética han permitido realizar cambios más precisos en el ADN y modificar las características de las células a placer. Sin embargo, hay grupos que se encargan de tratar de diseñar los microorganismos desde cero. Para estos grupos de investigación, un genoma totalmente sintético y diseñado racionalmente promete oportunidades de control sin precedentes de la función celular. Con estos organismos, por tanto, se podrían producir compuestos con interés industrial en biorreactores de una forma mucho más eficiente a la actual.
Hoy en día sigue siendo extraordinariamente complejo diseñar el genoma de un organismo. En 2019, la universidad ETH de Zurich trató de imitar y mejorar los resultados obtenidos con JCVI-syn3.0, y para ello modificar por ordenador el genoma completo de un organismo denominado Caulobacter crescentus, una bacteria de agua dulce con 6290 genes y 785.701 pares de bases. Tras sustituir 133.313 pares de bases, resultado fue una nueva bacteria denominada Caulobacter ethensis-2.0 con únicamente 799 genes y que, en teoría, era viable. Lamentablemente a fecha de hoy todavía no han podido introducirlo satisfactoriamente en un microorganismo.
Un salto evolutivo más
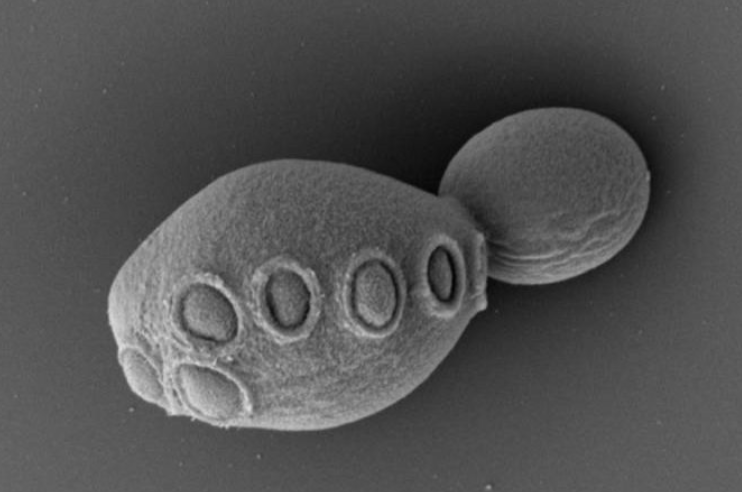
Por estas razones, 9 artículos publicados en la revista Cell Genomics han causado un revuelo en la comunidad científica. Tras 15 años de intenso trabajo, centros de investigación de Reino Unido, Estados Unidos, China, Singapur, Francia y Australia han recreado de forma sintética los 16 cromosomas que conforman el genoma de la conocida levadura Saccharomyces cerevisiae. Además de ello, han conseguido introducir 7,5 de estos cromosomas sintéticos de vuelta a la levadura con éxito. Como el ADN no está repartido de forma equitativa por los cromosomas, más de un 50% del genoma de la levadura resultante es artificial. Se trata de la primera vez que se consigue modificar con éxito un porcentaje tan elevado de una célula eucariota, mucho más compleja que cualquier bacteria.
Emplear S. cerevisiae no es fruto del azar, hace miles de años que la humanidad utiliza esta levadura para la producción de pan y bebidas fermentadas, así como otros compuestos químicos. Por ello, comprender y aprender a controlar todas las características de su genoma a ese nivel puede ayudar a obtener nuevos productos alimenticios, químicos y farmacológicos.
Levadura 2.0
El nombre de esta nueva levadura es Sc2.0 y presenta una serie de características muy interesantes y nunca vistas anteriormente en ningún organismo. Entre estas características, los investigadores han creado un cromosoma especial. En él se agrupan todos los genes que codifican para el ARN de transferencia, una molécula necesaria para la producción de proteínas en la célula. Normalmente, estos genes se encuentran repartidos por todos los cromosomas, pero los investigadores creen que agrupándolos todos en el mismo pueden aumentar la eficiencia de la levadura.
Además, han conseguido introducir un sistema dentro del genoma al que han denominado SCRaMbLE, que permite que los genes se mezcles y se reorganicen. De este modo se pueden obtener poblaciones de levadura distintas y crear una mayor diversidad de forma controlada.
Para insertar los cromosomas artificiales en las levaduras comenzaron cambiando únicamente uno de los cromosomas. Es decir, crearon 16 cepas de levadura distintas, cada una con 15 cromosomas naturales y uno sintético, distinto en cada una de las cepas. Tras realizar una serie de ajustes y comprobar que funcionaban correctamente, las dejaron reproducirse y fueron tomando las levaduras que tenían una mayor cantidad de cromosomas sintéticos. De este modo, obtuvieron levaduras con un número creciente de cromosomas sintéticos dentro de ellas. Para aumentar la eficiencia del método, crearon un sistema llamado “substitución cromosómica” con el que consiguieron transferir 7,5 cromosomas a una de las levaduras.
El próximo reto es acabar de incluir el resto de los cromosomas sintéticos a la levadura y crear, así, el primer organismo eucariota 100% sintético. Aunque ese objetivo sea el fin del proyecto, los investigadores afirman que se trata más bien de un principio. Con un genoma completamente sintético y modificable se puede conseguir que la levadura produzca prácticamente cualquier molécula de interés desde fármacos a bioplásticos o biocombustibles.
nationalgeographic.com.es
Fuente: https://formato7.com/2023/11/16/crean-una-bacteria-artificial-que-podria-ser-el-futuro-de-la-biotecnologia/
